Possible Molecular Basis behind the late surge of COVID-19 in Algeria.
الأسس الجزيئية المحتملة وراء الإرتفاع الأخير لحالات كوفيد-19 في الجزائر
الأسس الجزيئية المحتملة وراء الإرتفاع الأخير لحالات كوفيد-19 في الجزائر
Algeria witnesses a visible surge in the rise of COVID-19 cases and hence a quite worrying spread of the SARS-CoV-2 viruses. It's obvious that a set of objective causes are behind the rise including low awareness level of the danger the virus constitutes, lack of respect to the preventive measures such as social distancing, mask wearing and negligence .. etc.
تشهد الجزائر إرتفاعا ملحوظا في حالات الإصابة بمرض كوفيد-19 وبالتالي إنتشار مرعب لفيروس سارس-كوف-2. من الواضح أن هناك أسباب موضوعية لهذا الإرتفاع تقع مسؤوليته الكبرى على قلة وعي الناس بخطورة الفيروس وعدم احترامهم لإجراءات الوقاية مثل التباعد الجسدي، إرتداء القناع والإستهتار .. الخ
In addition to the human factor, there seem to be other causes beyond the human activity related to the nature of mutations acquired by the SARS-CoV-2 virus evolving on the Algerian scene.
بالإضافة إلى دور العامل البشري، يبدو أن هناك أسباب خارجة عن إرادة النشاط البشري تعود إلى طبيعة الطفرات التي تشكلت في جينوم الفيروس المتطور على الساحة الجزائرية.
A large number of Algerian local genome sequences from the many COVID-19 infected regions would help in constructing detailed mutability map and better assessment for potential cures discovery and even identification of genomic regions to be vector-based PCR amplified for local vaccine(s) production.
كان من شان القيام بالتعرف على تسلسل الجينوم الفيروسي في سلالات سارس-كوف-2 من عديد المناطق المصابة في الجزائر أن يوفر فهما دقيقا لخارطة التحورات الجينومية للفيروس والتي من شأنها إنارة الطريق إلى السبل المثلى لمجابهة الفيروس وحتى التعرف على الأجزاء المهمة في جينوم الفيروس المحلي والتي يمكن أن تنفع في إنتاج لقاح أو خليط من اللقاحات الفاعلة بتقنيات التكثيف بالزرع-الجيني والبلمرة .
However, the situation is that Institute Pasteur Algeria has made available only 3 genomic sequences of SARS-CoV-2 genome isolated from Blida province patients; Two (2) from Boufarik and one (1) from the city of Blida which are available via the 'Viruses' database (https://www.bioinformaticstools.org/viruses/) and which have been written about in the post entitled " Algerian Complete Genome sequences of SARS-CoV-2 strains detected in Blida province "
available via the link: https://www.facebook.com/bioinformaticstools/posts/3056044324450784
ولكن واقع الحال أن معهد باستور الجزائري قام بإنجاز ثلاث سلاسل جينومية فقط والني تم عزلها من الإصابات بولاية بليدة؛ سلسلتين (2) من منطقة بوفاريك و واحدة (1) من مدينة البليدة والمتوفرة من خلال قاعدة_بيانات "فيروسات" على الموقع (https://bioinformaticstools.org/viruses/) والتي تم التعرض إليها في منشور سابق (" الجزائر؛ سلاسل الجينوم الكامل لفيروس كورونا سارس-كوف-2 المكتشف في ولاية البليدة .. " على الرابط : https://www.facebook.com/bioinformaticstools/posts/3056044324450784).
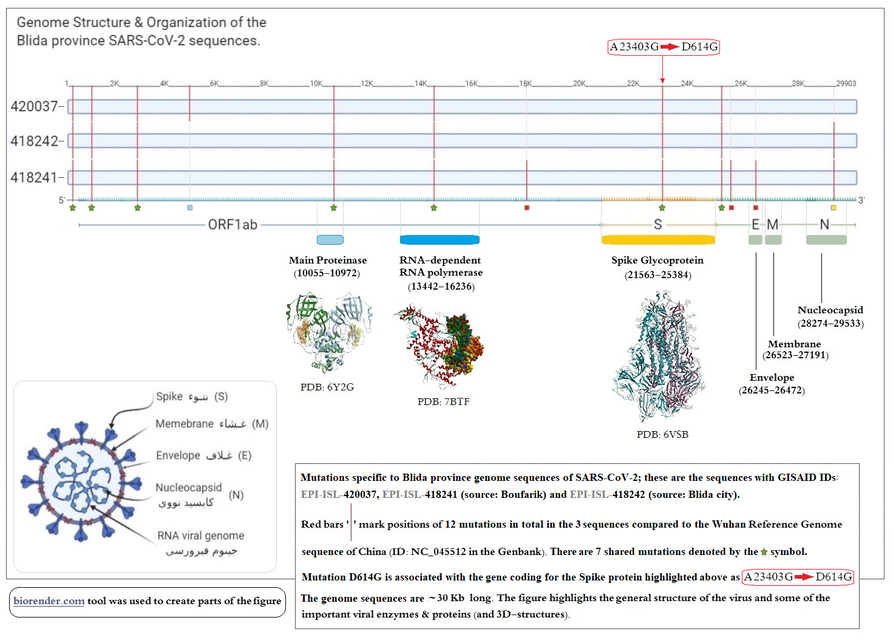
Multiple Sequence Alignment study, by the SB&B[*] group, of these 3 genome sequences (local versions of SARS-CoV-2) together with the reference genome of Wuhan - China, revealed 12 mutations in local strains among which 7 are shared between the sequences (denoted with a little green star in image 1 attached below). Multiple Sequence Alignment study, by the SB&B[*] group, of these 3 genome sequences (local versions of SARS-CoV-2) together with the reference genome of Wuhan - China, revealed 12 mutations in local strains among which 7 are shared between the sequences (denoted with a little green star in image 1 attached below).
دراسة و تحليل مقارنة السلاسل، من إنجاز فريق البحث، أنظر[*]، لهذه السلاسل الثلاث فيما بينها ومع سلسلة الجينوم المرجعي من ووهان-الصين، أوضحت أن سلاسل الجينوم المحلية تشتمل على 12 طفرة منها 7 طفرات مشتركة بين السلاسل المحلية موضحة في الصورة الأولى 1 أدناه برمز النجمة (خضراء اللون).
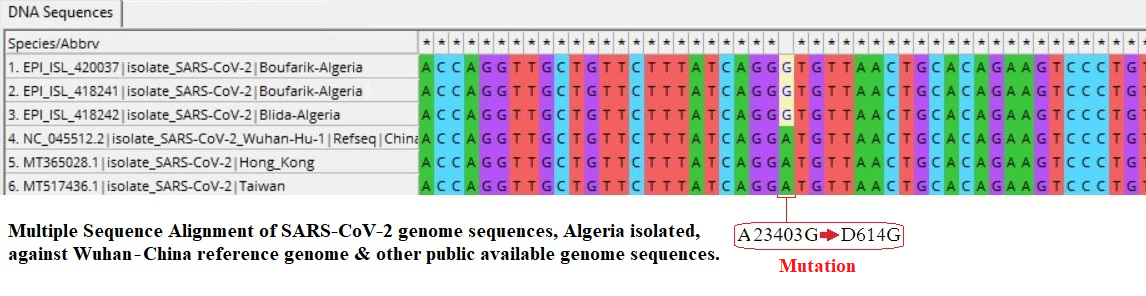
This post focuses on one of the 7 shared mutations; that is the mutation located on the viral Spike protein used by SARS-CoV-2 to bind the human ACE2 receptor and enter the infected cells. This mutation occurs at the nucleotide base number 23403 where the base Adenine (A) mutated into Guanine (G) (that is A23403G), see the alignment partially depicted in image 2.
The mutation resulted in replacing the amino acid number 614 Aspartic (Asp or D) by Glycine (Gly or G) in the translated product protein - the Spike protein; hence the notation 'A23403G -> D614G ' red-boxed in image 1.
هذا المنشور يركز على إحدى الطفرات السبعة المشتركة؛ وهي الطفرة الواقعة على مستوى البروتين الفيروسي الشوكي، الذي يستخدمه الفيروس سارس-كوف-2 ليرتبط بالمُسْتَـقْبِل البشري 'آيس-2' ويدخل عبره الخلايا المصابة. هذه الطفرة تتمثل في استبدال قاعدة النيوكليوتبد رقم 23403 من النيوكليوتيد 'أدنين' إلى 'الغوانين'، أنظر مقارنة السلاسل المعروضة جزئيا في الصورة 2.
تسببت هذه الطفرة في تغير الحمض الأميني 614 من 'الأسبرتيك' إلى 'الجليسين' في البروتين المترجم عن المورثة و هو البروتين الشوكي؛ هذا تبيان الوسم الذي يصف هذه الطفرة:
' D614G <- A23403G '
المؤطرة بالعلبة-الحمراء في الصورة 1.
Structural & functional effects of the D614G mutation on the Spike protein in Algeria's local SARS-CoV-2 [*]:
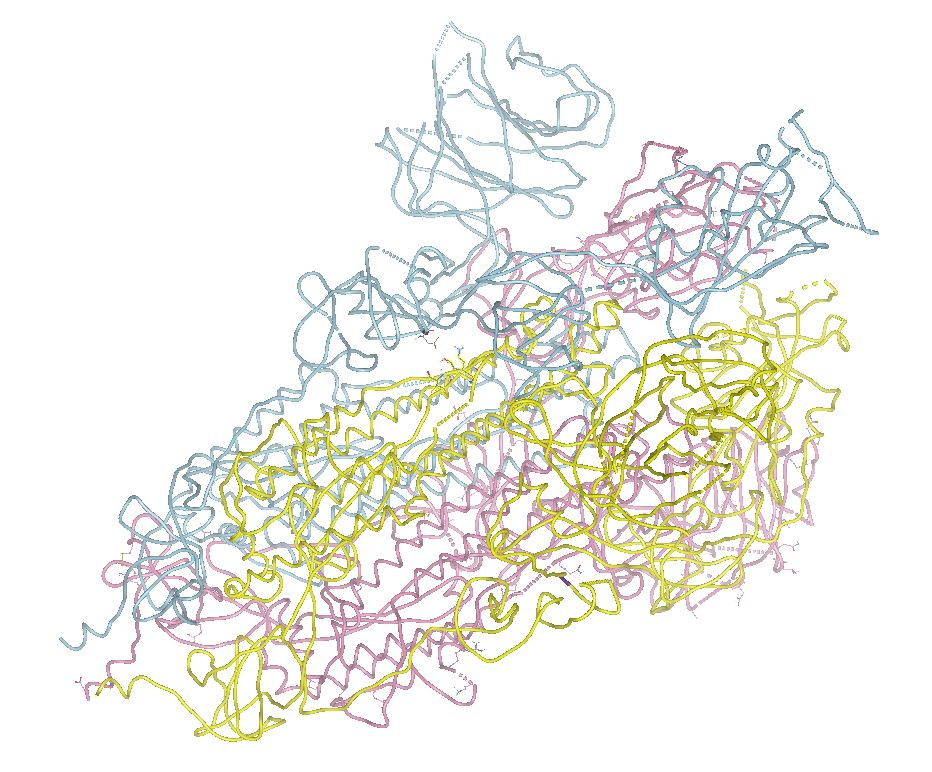
This study has used the available structure of the SARS-CoV-2 Spike protein bearing the PDB id '6vsb', see [#], as a starting model; the Spike protein is a trimer, see images 3 and 4, made of three chains a (bluish-grey), b (in yellow) and c (in pink) .
D or Aspartic is an acidic amino acid, bears a net negative charge (-), hydrophilic and is quite bulky compared to G or Glycine which is small, nonpolar and lightly hydrophobic. This means that this mutation is a big change in the structural, chemical and functional properties at the localized region where it happened in the protein.
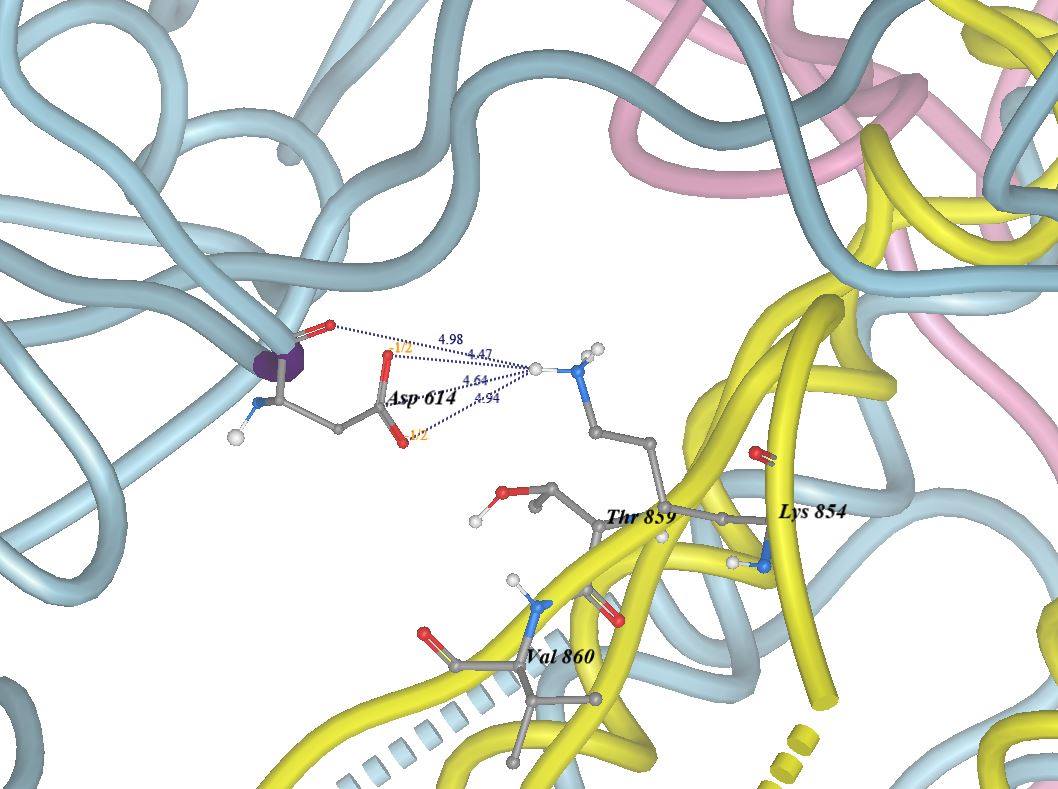
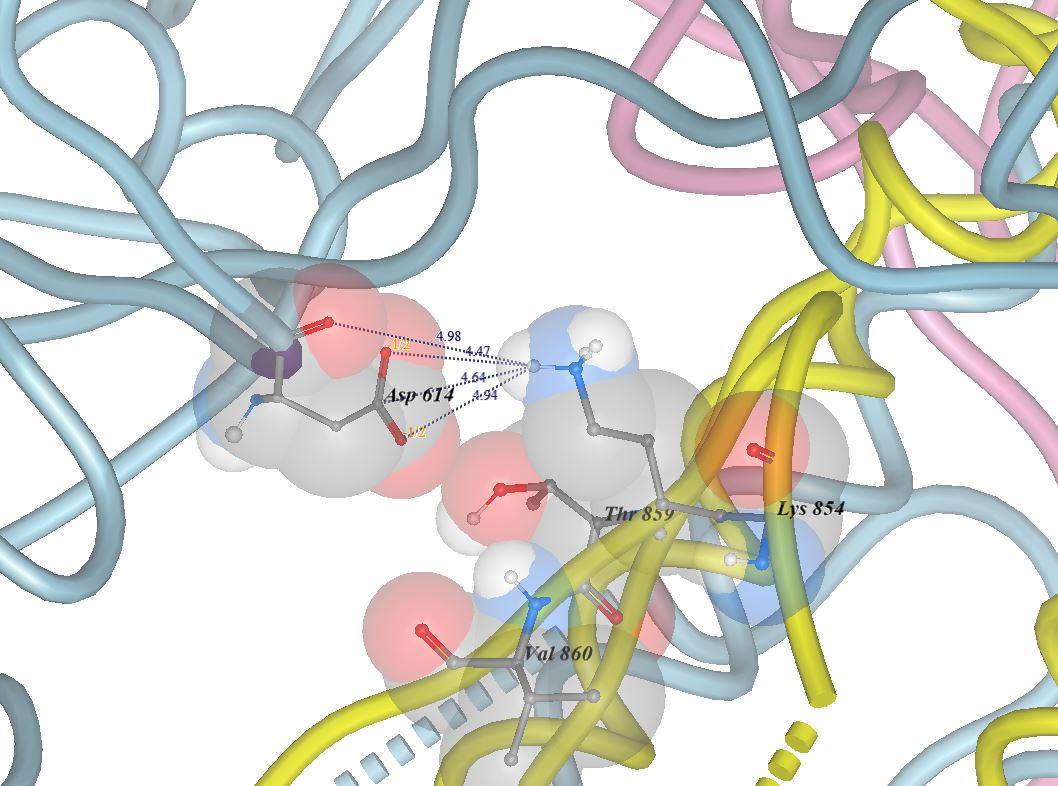
In the wild-type Wuhan SARS-CoV-2 virus, the D-614 makes an extensive inter-chains network of bonds between the 3 chains constituting the Spike protein; D-614 from each chain (e.g. chain a) forms good structure stabilizing contacts with amino acids Lysine-854 (Lys or K), Threonine-859 (Thr or T) and Valine-860 (Val or V) all of which found in each of the adjacent chains (e.g. chain b), see images 5 & 6.
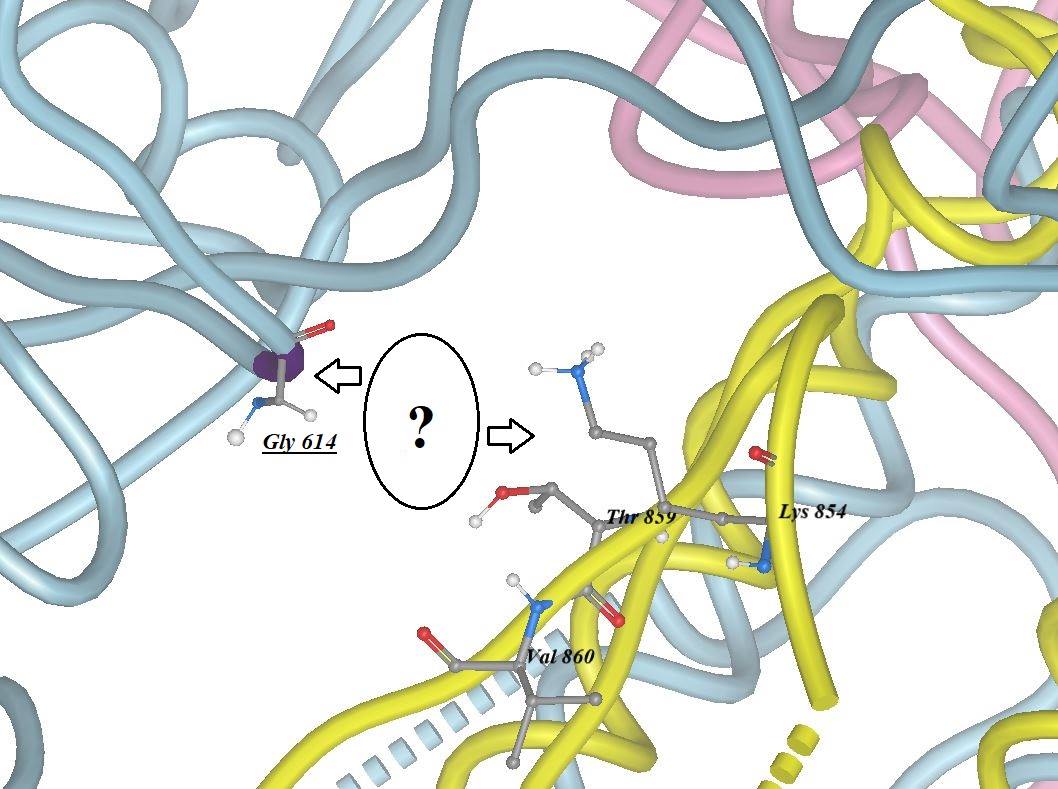
The replacement of D-614 with G-614 in the mutant-type Spike protein (Algeria's virus), costs the protein to loose this particular structure stabilizing inter-chains contacts (described above) and corresponding structural/functional effects would result in the Spike protein, see image 7.
In this case it seems that such a mutation, D614G , has resulted in an "enhancement towards a better binding between the viral Spike protein and the human ACE2 receptor" and hence in a "potentially faster/wider spread capability of the infection with a greater rate compared to the original virus of Wuhan".
The rational behind this prediction of the functional effect by this mutation is based on the fact that the mutant virus has shown wider spread compared to China and on the fact that this mutation has been found common to most SARS-CoV-2 genomes sequenced post China infection period.
التأثيرات التركيبية والوظيفية للطفرة
D614G
على البروتين الشوكي للفيروس سارس-كوف-2 المحلي [*]:
في هذه الدراسة تم استخدم التركيب الفراغي للبروتين الشوكي الموسوم بـ
'6vsb'
، من قاعدة_البيانات المتخصصة في التراكيب الفراغية
PDB
، أنظر النقطة [#]، كنموذج ابتدائي؛ البروتين الشوكي لهذا الفيروس هو ثلاثي الجدائل حيث يتألف من ثلاثة سلاسل a (باللون أزرق-رمادي)، b (باللون الأصفر) و c (باللون الوردي) أنظر الصور 3 (التركيب العام للفيروس) و 4 (البروتين الشوكي).
الحمض الأميني 'أسبارتيك' له صفة حامضية مرتفعة، يحمل شحنة كلية سالبة (-)، محب للماء وذا حجم كبير بالمقارنة مع 'الجليسين' صغير الحجم، غير مشحون و كاره للماء نسبيا. هذا يعني أن هذه الطفرة تمثل تغيرا كبيرا في الصفات التركيبية، الكيميائية و الوظيفية للمنظقة المحددة من البروتين حيث وقعت الظفرة ..
بالنسبة إلى فيروس ووهان سارس-كوف-2 النوع_الوحشي
wild-type
، أي الغير متطفر، فإن الحمض الأميني 'أسبارتيك' - 614 يقوم بشبكة من الروابط بين_السلاسل المؤلفة للبروتين الشوكي. يقوم الحمض الأميني 'أسبارتيك' - 614 من كل سلسلة (مثلا السلسلة a) بروابط مُـثَـبِّـتة للتركيب الفراغي الكُلِّي مع الأحماض الأمينية: 'ليسين' رقم 854، 'ثريونين' رقم 859 و 'فالين' رقم 860 المتواجدة في السلسلة المحاذية (مثلا السلسلة b)، أنظر الصور 5 و 6.
عملية تغيير الحمض 'أسبارتيك' - 614 بـالحمض 'جليسين'-614 في جينوم الفيروس المتطفر
mutant-type
المحلي (جينومات منطقة البلبدة) تجعل البروتين الشوكي يفقد شبكة الروابط المُـثَـبِّـتة هذه بين سلاسل البروتين الشوكي (الموصوفة أعلاه) والتي يتحتم أن ينجم عنها تأثير تركيبي ووظيفي مقابل، أنظر الصورة 7.
في هذه الحالة تحت الدراسة، يبدو أن الطفرة 'أسبارتيك' - 614 -'جليسين'
D614G
تسببت في " تعزيز إمكانية الإرتباط بين البروتين الشوكي والمستقبل البشري 'آيس-2' " وبالتالي ساهمت في تمكين الفيروس من " الإنتشارالسريع و بمعدل أكبر من إنتشار الفيروس الأصلي الآتي من ووهان
Wuhan".
التخمين المنظقي وراء هذا التقدير أو التوقعات الجزيئية الأولية وراء التأثيرات الوظيفية للطفرة 'أسبارتيك' - 614 -'جليسين'، الموصوفة أعلاه، تعتمد على حقيقة أن ظاهرة الإنتشارالسريع للفيروس بالمعدل الحالي شوهدت 'خارج الصين' كما أن هذه الطفرة تم التعرف عليها في العديد من الدراسات بأنها 'الأكثر شيوعا' في حوالي 75% من سلاسل جينومات سارس-كوف-2 ما بعد فترة الإصابات في الصين.
More structural study is planned to confirm the relation between the speed of spread of COVID-19 and the D614G mutation and answering the question mark in image 7.
Nature and details of the other mutations characterizing the local Blida province sequences of SARS-CoV-2 genome, shown in image 1, are to come back to in future investigations.
من المخطط إجراء المزيد من الدراسات التركيبية لتأكيد العلاقة بين سرعة إنتاشار مرض كوفيد-19 والطفرة 'أسبارتيك' - 614 -'جليسين' وللإجابة على السؤال من وراء علامة الإستفهام موضوع الصورة 7.
طبيعة وتفاصيل الطفرات الأخرى المميزة لـ سلاسل جينوم الفيروس سارس-كوف-2 المحلية، المشار إليها في الصورة 1، هي موضوع دراسات مستقبلية.
___________________________________
[*] SB&B: the studies (Sequence Alignment and Structural & Functional Effects) are done by the SB&B group: Structural Biology & Bioinformatics group, Dept. of Biology, University of Saida.
الدراسات الأولية المشار إليها أعلاه (مقارنة السلاسل ودراسة التأثيرات الفراغية والوظيفية) قام بها فريق البحث للبيولوجيا التركيبية/الفراغية و المعلوماتية_الحيوية، قسم البيولوجيا، جامعة سعيدة
[#] Explore the Spike protein structure '6vsb' at the SSFS tool (by SB&B group):
يمكن استكشاف التركيب الفراغي للبروتين الشوكي '6vsb' باستخدام التطبيق SSFS (من إنتاج فريق البحث SB&B) على الرابط:
See also the relevant post:
أنظر المنشور ذا العلاقة:
___________________________________
See also the following site:
أنظر كذلك الموقع التالي:
Best of the New Year regards
أزكى تحيات العام الجديد
Author: A. Rachedi
E-mail: rachedi@bioinformaticstools.org
Date: - 25 December 2021
- Facebook vr.1 POST ID=3152199394835276 - 28 July 2020
E-mail: rachedi@bioinformaticstools.org
Date: - 25 December 2021
- Facebook vr.1 POST ID=3152199394835276 - 28 July 2020
Images: