The new SASR-CoV-2 variants .. VUI-202012/01 & 501Y.V2 .. السّلالات الجديدة لفيروس كرورنا
& the high transmissibility phenomena .. . .. . .. .. . .. . .. وظاهرة الإنتشار السريع
نحو تفسير جُزَيْئِي محتمل لزيادة سرعة إنتشار السّلالات الجديدة لفيروس كورونا؛ VUI-202012/01 و 501Y.V2 و إرتفاع الإصابات بـ كوفيد-19 في بريطانيا\أوروبا و جنوب إفريقيا ..
Towards a molecular based explanation of the high transmissibility of the new variants of the corona-virus; VUI-202012/01 & 501Y.V2 and the rise of COVID-19 cases in England/Europe and South Africa ..
في تقديراتنا، ظهور المتحور VUI-202012/01[*] في جنوب شرق إنجلترا و انتقاله إلى الكثير من دول أوروبا يُشَكِّلَ الموجة الثالثة لمرض كوفيد-19 في قارة أوروبا و أما المتحور 501Y.V2 فقد تسبب في الموجة الثانية بالنسبة لجنوب إفريقيا. كلا المتحورين يتمتعان بالمقدرة الكبيرة على الإنتشار السريع كما وجد أنها قادرة على إصابة الشباب بكفاءة أكبر من المتحور السابق؛ انظر المنشورين السابقين:
In our judgement, while the advent of the new 501Y.V2 variant of the SARS-CoV-2 virus represented the second wave of Covid-19 disease in South Africa, the newly evolved VUI-202012/01[*] variant in the south east of England marked the third wave of the disease in Britain and then in Europe after the spread. Both new strains have been associated with a high level of spreading and have shown ability to infect young adults. See the previous post in the subject:
- [*] هذا المتحور، VUI-202012/01، أصبحت تصفه الجهات البريطانية الرسمية الآن بـ VOC-202012/01 أي 'Variant of Concern, year 2020, month 12, variant 01' أو 'متحور مثير_للقلق، سنة 2020، الشهر 12، المتغير 01'
The variant 'VUI-202012/01' has been renamed by the Public Health England to be 'VOC-202012/01' which stands for 'Variant of Concern, year 2020, month 12, variant 01'
للتذكير، فإن المتحور السائد و الذي سبق ظهور كلا من VOC-202012/01 و 501Y.V2 هو كذلك متطفرة و تعرف بـمتحورة D614G أو variant D614G والتي نتجت بعد إنتقال العدوى الفيروسية، أو الفيروس SARS-CoV-2 الأصلي المرجعي [&]، من مدينة يوهان Wuhan إلى خارج الصين . تميز المتحور D614G بالسرعة في الإنتشار و العدوى و إن كانت أقل سرعة من المتحورين موضوع الإهتمام في هذا المنشور. في المقابل، فإن المتحور D614G أظهر حملاً فيروسيًا منخفضًا ولم يُعرف عنه أنه يصيب الشباب بشكل ملحوظ.
It's important to keep in mind that the globally dominant coronavirus strain, the one before the new variants VOC-202012/01 and 501Y.V2, is also a variant/mutant that have been technically know as the D614G variant evolved out of the original/reference SARS-CoV-2 strain before it left Wuhan / China. The D614G strain also showed faster transmission though slower compared to the new variants. In contrast, the D614G strain showed low viral load and has not been known to noticeably infect young adults.
- [&] يمكن استكشاف جينوم الفيروس SARS-CoV-2 المرجعي و سلاسل البروتينات/الإنزيمات من قاعدة البيانات Viruses، فريق المعلوماتية_الحيوية والبيولوجيا الفراغية، قسم البيولوجيا، جامعة سعيدة، على الرابط أدناه:
The reference genome of the SARS-CoV-2 and related proteins/enzymes can be explored from the 'Viruses' database by the Structural Biology and Bioinformatics group, Department of Biology, University of Saida, via the link:
المزيد من التفاصيل حول السلسلة الجينومية المرجعية يمكن استكشفها إنطلاقا من الرابط المباشر لقاعدة البيانات Genbank على الرابط:
More details abut the refrence genome are available from the NCBI/Genbank site:
ما يلي محاولة لتفسير جوانب من العوامل الجُزَيئِية وراء شدة العدوى وسرعة إنتشار السّلالات الجديدة حيث ينبغي الأخذ في الإعتبار النقاط التالية:
The following is an attempt to provide an explanation to molecular basis behind the transmissibilty speed of the viruses:
1. هناك تأثير تراكمي تعزيزي للطفرات الفاعلة\المعتمدة من طرف الفيروس ذلك لأن كلا السلالتين الجديدتين VOC-202012/01 و 501Y.V2 تحتويان الطفرة D614G التي أظهرت سرعة و شدة في العدوى بالمقارنة مع الفيروس الأصلي/المرجعي، هذا التراكم من الطفرات "مسرعة النشر" هي ما أدى إلى زيادةٍ أكبر في سرعة الإنتشار و التي تم تسجيلها مؤخرا في بريطانيا وعموم أوروبا و كذلك جنوب إفريقيا.
1. There's a renforcing cumulative effect to the adopted mutations by the virus. This is because bof the new varaints VOC-202012/01 and 501Y.V2 already carry the mutation D614G which showed a faster transmissibilty of the virus compared to the original/reference one; this accumulation of mutations of same effect, we believe, are responssible for the further gearing up of the speed of transmissibilty seen in these new variants showed up in Britain and South Africa.
أنظر المنشور حول الأساس الجزيئي المحتمل للطفرة D614G:
أنظر كذلك المنشور حول عوامل أخرى محتملة مرتبطة بمستقبلات خاصة يبدو أن لها مساهمة فاعلة في سرعة الإنتشار للفيروس:
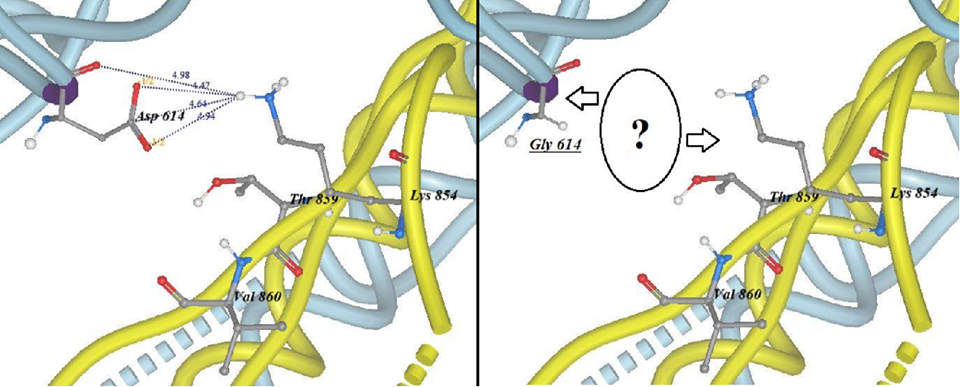
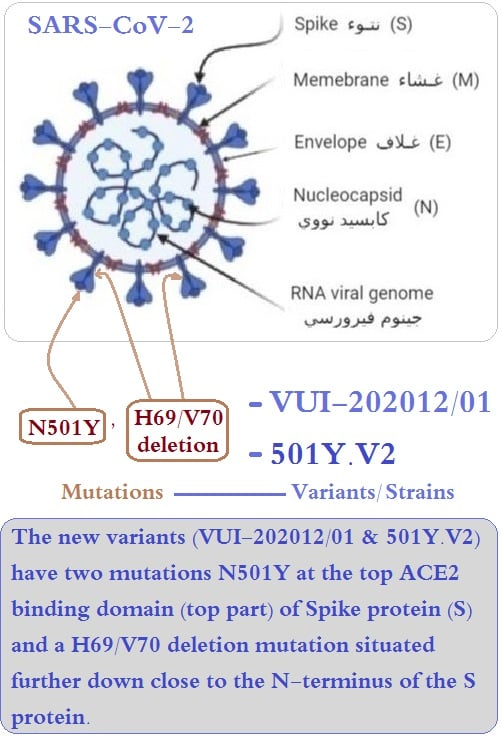
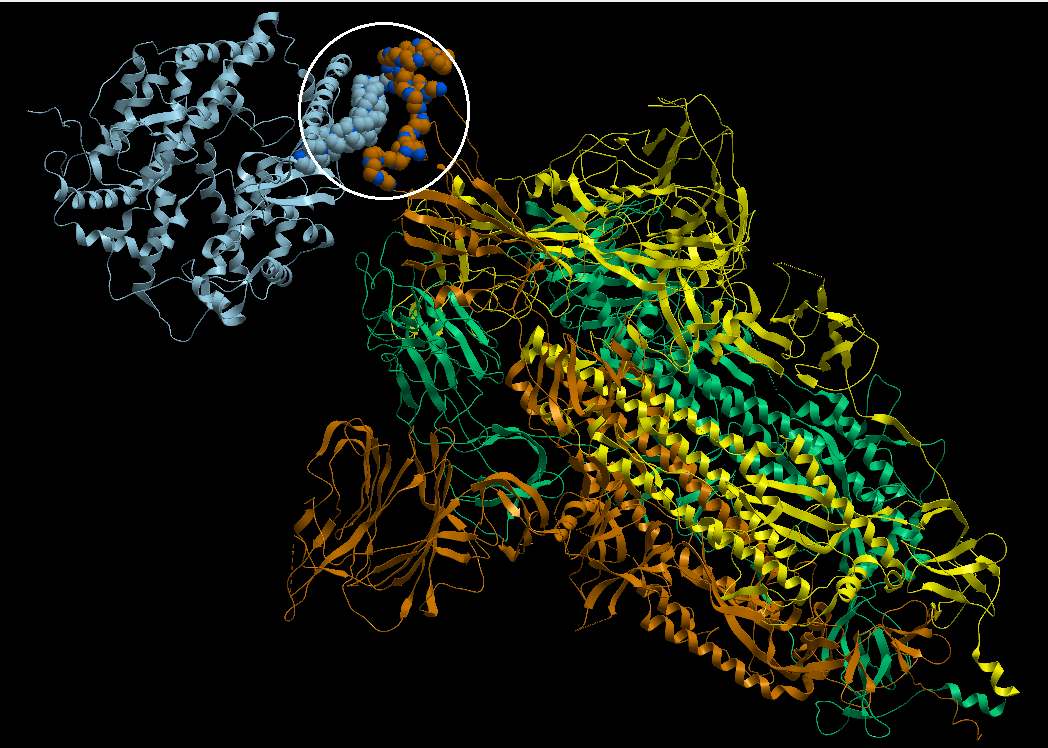
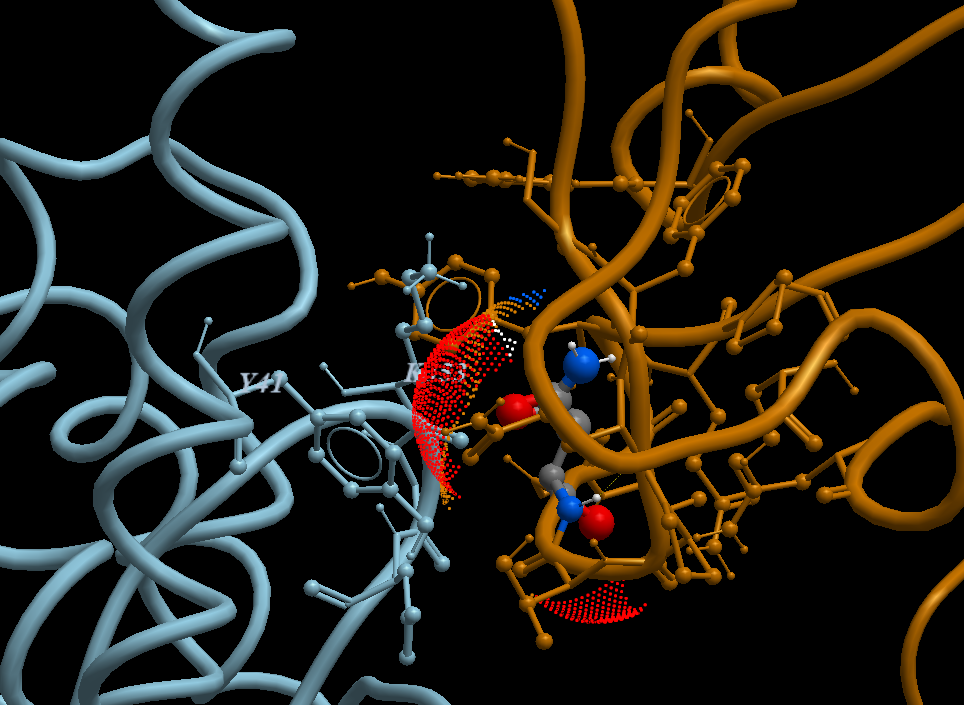
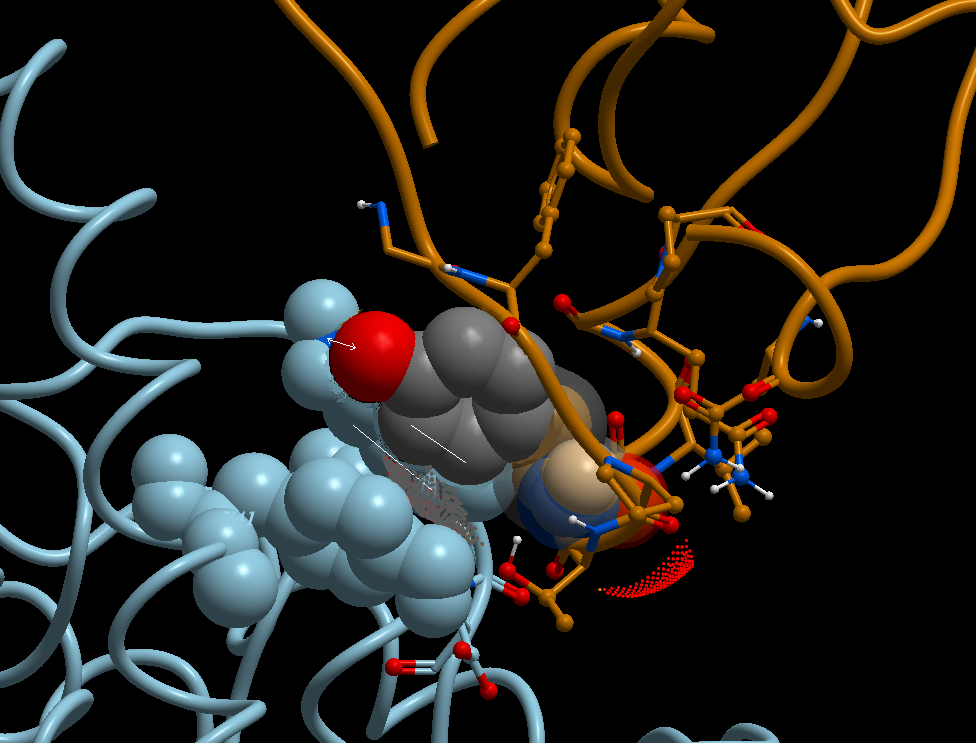
2. بالنسبة للمتحورين VOC-202012/01 و 501Y.V2 هناك طفرتين مشتركتين بينهما، أنظر الصورة 3 image، أي الطفرة N501Y و 'طفرة الحذف H69/V70' وهما كما يلي :
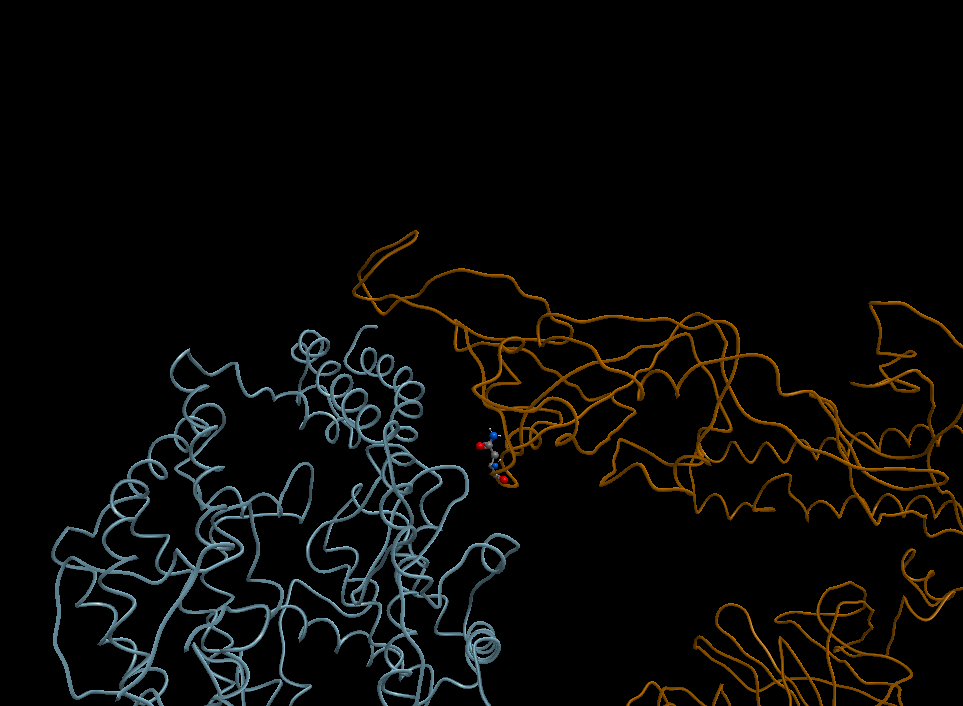
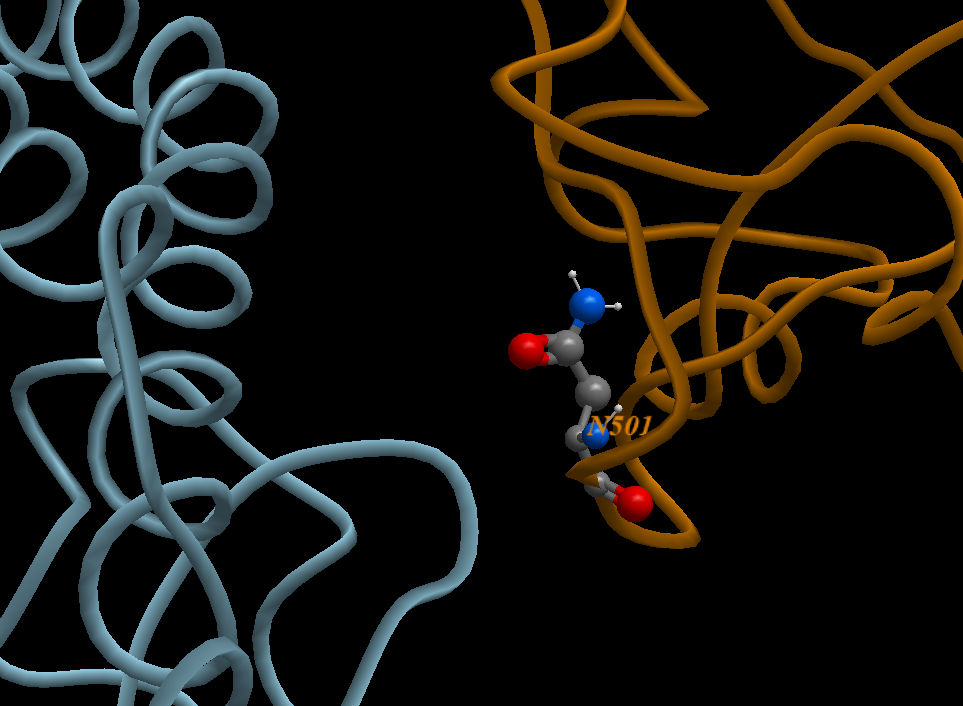
أ. الطفرة N501Y : حدث الطفر الجيني على مستوى الجين المسؤول عن إنتاج البروتين الشوكي Spike protein و تتمثل في تحول الحمض الأميني أسبرجين Asparagine أو ASN أو N إلى الأحمض الأميني تيروسين Tyrosine أو TYR أو Y في الموقع 501 على طول سلسلة متعدد البيبتيد المؤلفة للبروتين الشوكي.
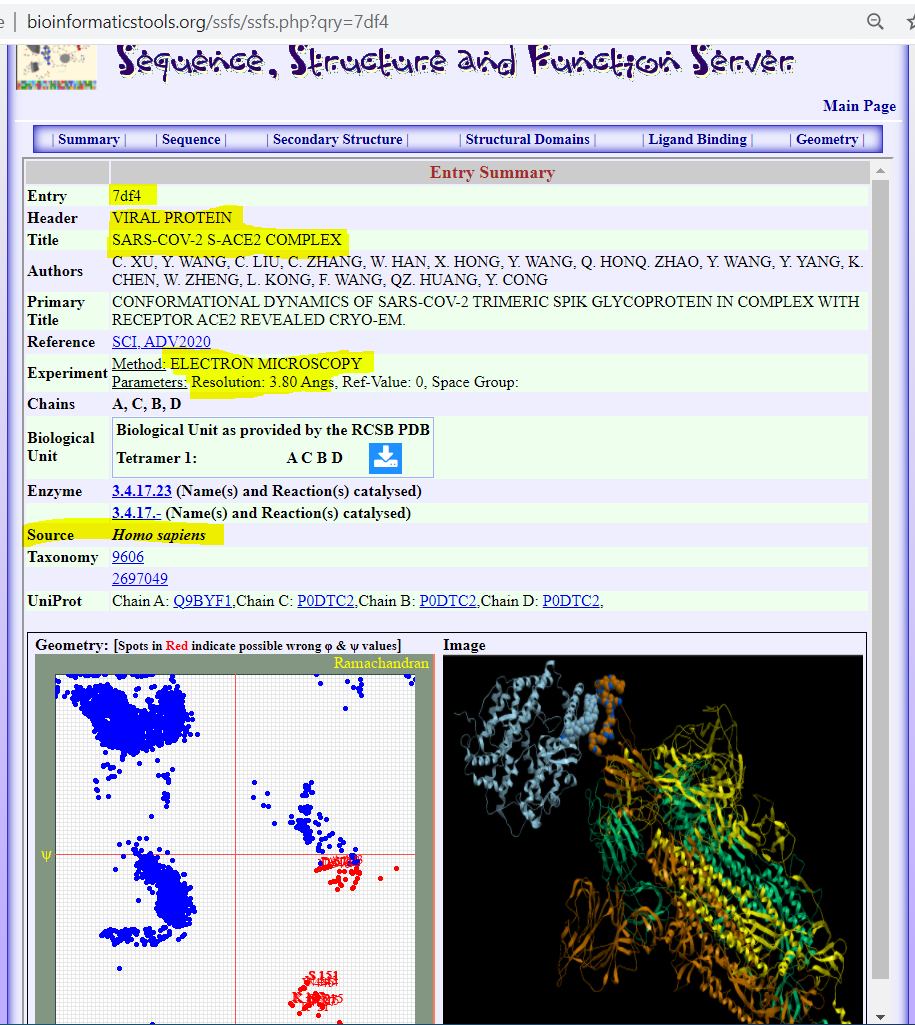
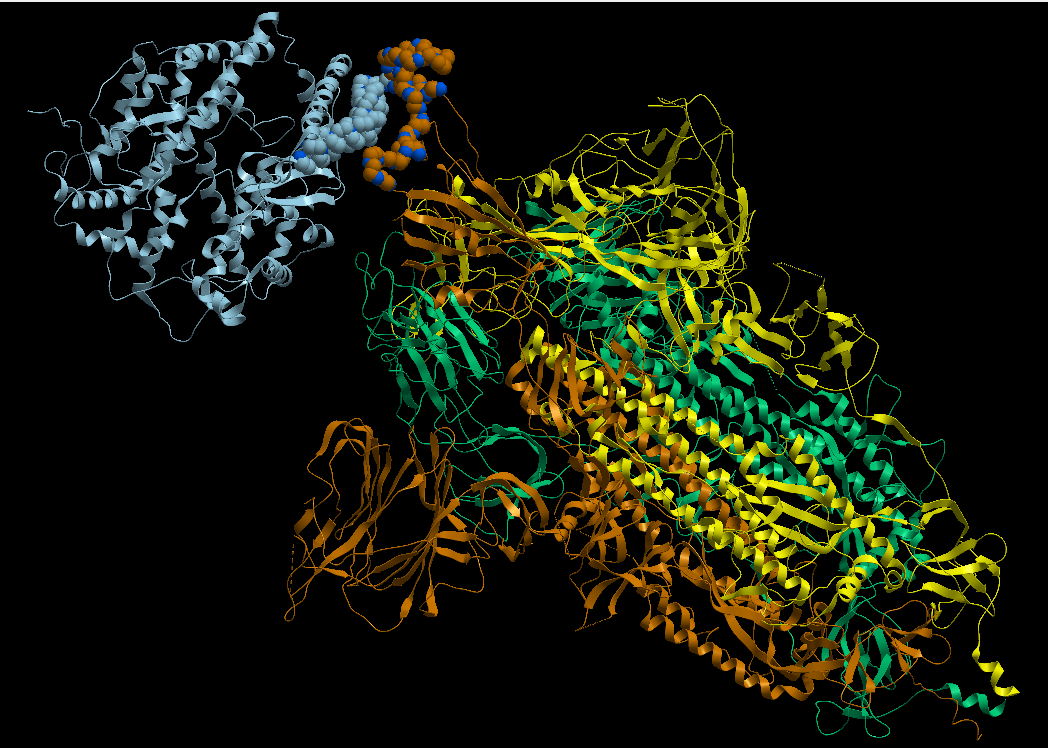
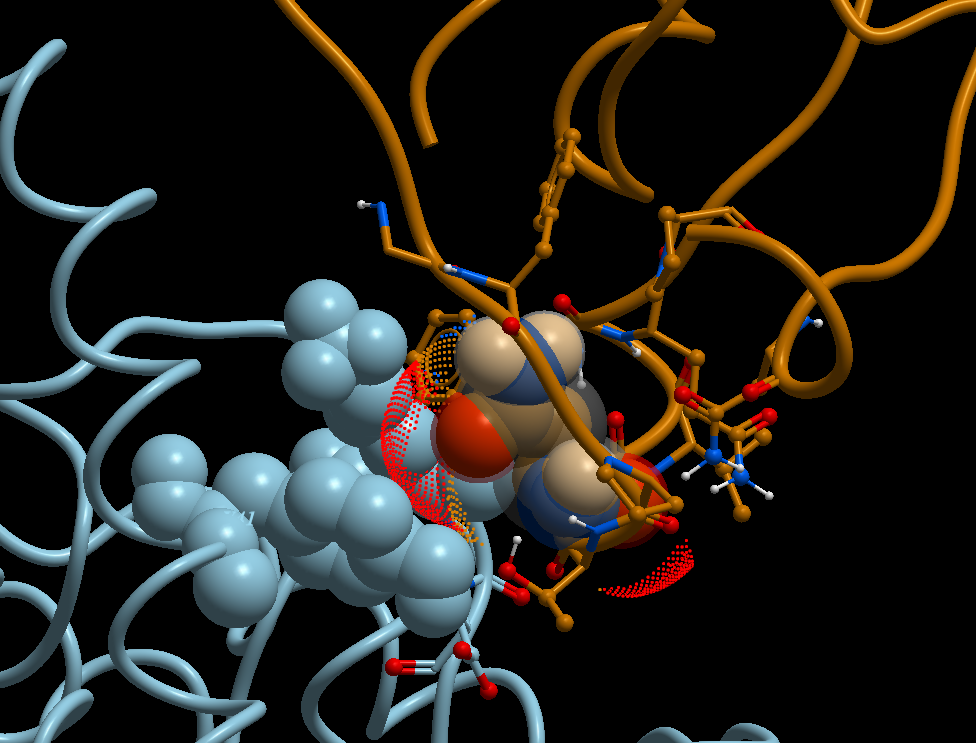
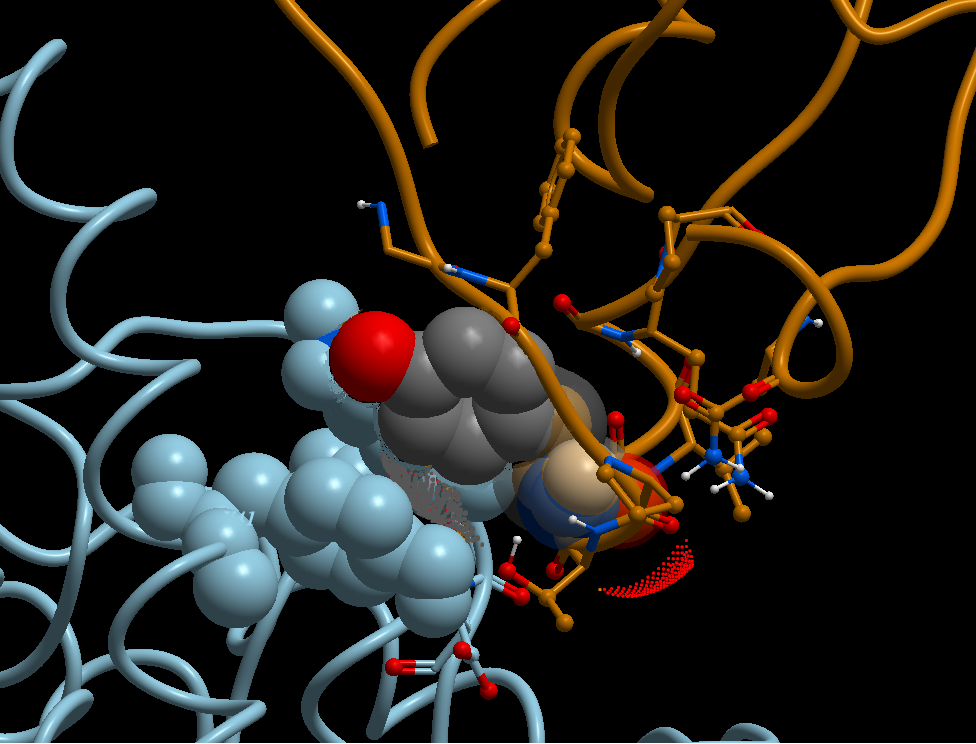
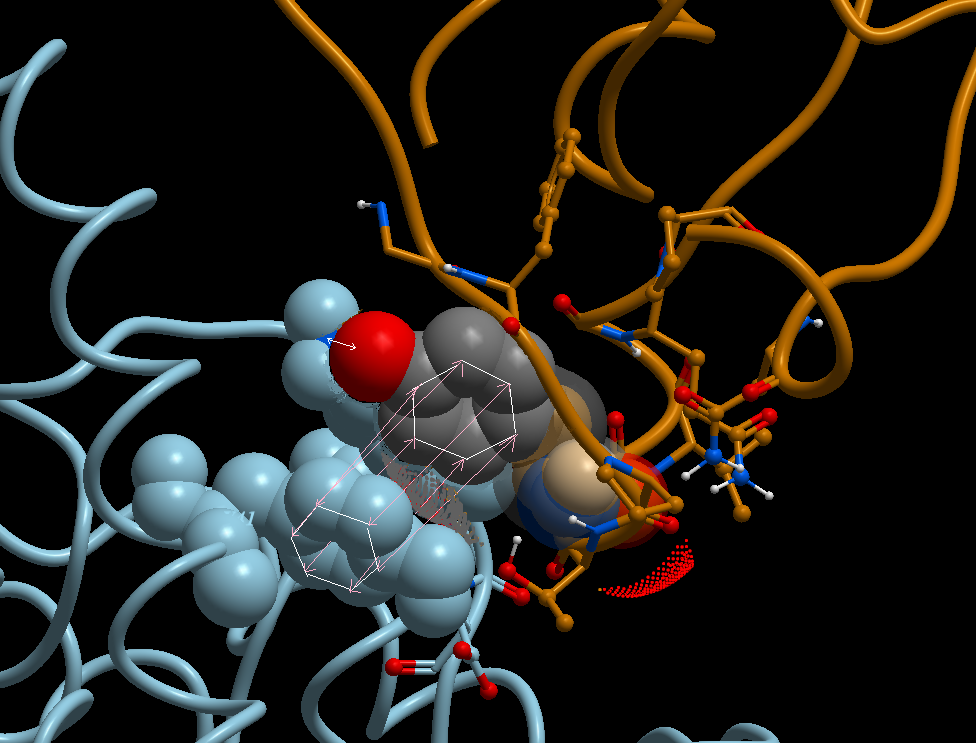
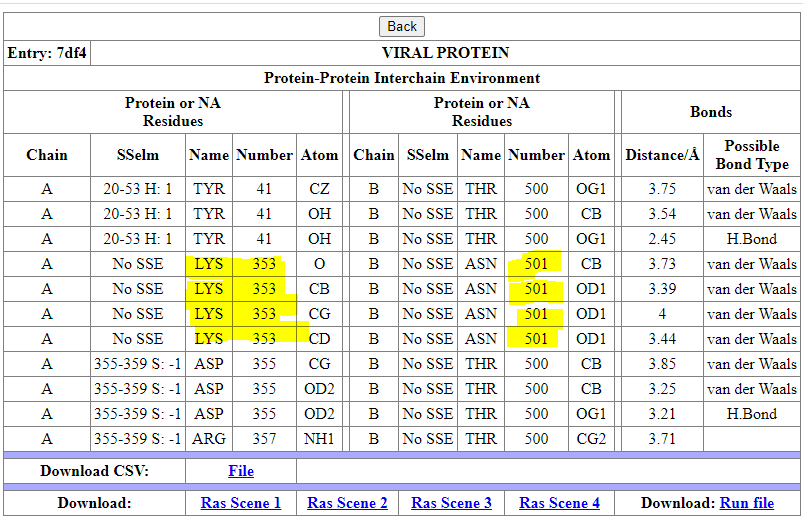
بناءا على النمدجة الجزيئية الأولية[%] من إنجاز فريق البحث "البيولوجيا الفراغية و المعلوماتية_الحيوية"[£.1]، فإن تغيير الفيروس للحمض الأميني أسبرجين Asparagine - ASN - N رقم 501 إلى حمض التيروسن Tyrosine - TYR - Y، أدى إلى زيادة نسبية في قوة الإرتباط بين البروتين الشوكي للفيروس و المستقيل ACE2، الصور 5 إلى 11a (images 5 to 11a).
- [%] تعتمد دراسة النمذجة الجزيئية هذه على التركيب الفراغي لمعقد البروتين الشوكي لفيروس كورونا SARS-CoV-2 في حالة إرتباط مع المستقبل ACE2 البشري Homo sapiens المتوفر من قاعدة البيانات PDB تحت الرمز/المدخل 7df4 كما هو موضح عبر التطبيق SSFS، الصور 4 و 5 (images 4 and 5)، من موقع المعلوماتية_الحيوية و البيولوجيا الفراغية [£.2].
- [£.1] و [£.2]: فريق البحث Structural Biology & Bioinformatics تابع لقسم البيولوجيا، جامعة سعيدة، الجزائر، على الموقع:
كما هو مبين في الصورة 11a image، من بين الصور المرفقة مع هذا المنشور، تتوقع الدراسة النمذجة أن استبدال الفيروس (المتطفر الجديد) للحمض الأميني أسبرجين N إلى تيروسين Y أدى إلى إنتاج إتصال أقوى بين الحمض الأميني في الموقع 501 البروتين من الشوكي للفيروس و منطقة الإتصال مع المستقبل ACE2 حيث يوجد الحمضين الأمينيين تيروسن Y رقم 41 و الليسين K رقم 353؛ أنظر التعليق المرافق للصورة. بينما في المقابل فإن الأسبرجين N رقم 501 في الفيروس (السابق) يقوم بالإرتباط بحمض أميني واحد فقط متواجد في البروتين ACE2 و هو الليسين K 353، انظر الصورة 12 image.
لفهم أوضح لموضوع النمذجة، الرجاء الرجوع إلى الصور 5 إلى 11a-c (images 5 to 11a-c) مع التعاليق المرفقة معها.
ملخص التقديرات الأولية لنمذجة الطفرة N501Y هو أنهذا التحول في كلا السلالتين، موضوع النقاش، أدى إلى تعزيز إضافي في قوة الإرتباط بين البروتين الشوكي للفيروس و مستقبلات ACE2 الأمر الذي من شأنه أن يفسر سرعة إنتقال العدوى high transmissibility of infection و إمكانية الإصابة بالعدوى بفعل تركيز فيروسي أقل high viral load و إصابة فئة الشباب infection of young adults وهي العوامل الثلاث المميزة لمرض كوفيد-19 في حلته الجديدة الناتجة عن السّلالات الحديثة.
ب. 'طفرة الحذف H69/V70' أو H69/V70 deletion والتي أدت إلى حذف الحمضين الأمينيين هيسدين 69 و فالين 70 من البروتين الشوكي.
هذه الطفرة ليس لها تأثير واضح على العوامل المذكورة أعلاه و لكن تؤثر على الجوانب الإكلينية المتعلقة بالتأثير على دقة الكشف عن الإصابة بفيروس كورونا المستجد الناتج عن السّلالات الحديثة .. الرجاء الروجع إلى المنشورات المرفقة أدناه الرقمة [1] و [2].
لعل تقديرات النمذجة المشار إليها أعلاه تمثل جزءا من الإجابة و لكنها بالتأكيد لا تمثل الإجابة الكاملة.
للمزيد، تابع المنشورات ذات العلاقة بالفيروس SARS-CoV-2 و مرض كوفيد-19 لهذه الصفحة على الروابط التالية:
For more, do refer also to the posts relevant to the SARS-CoV-2 virus and COVID-19 disease:
[1]. Strain '501Y.V2', another new Coronavirus Variant in South Africa then in England.
السلالة '501Y.V2' نوع متطفر جديد آخر لفيروس كورونا في جنوب إفريقيا ثم في إنجلترا.
[2]. سلالة جديدة VUI-202012/01 من فيروس كورونا تظهر في بريطانيا ..
VUI-202012/01 a new coronavirus variant identified in UK ..
[3]. The Nature of Pfizer and Moderna vaccines & how do they work ?
طبيعة لقاحي ' فايزر ' و 'مُودَارْنَا ' و كيف يعملان؟
[4]. "WHO recommends against the use of remdesivir in COVID-19 patients" .. what does it mean ?
نصائح منظمة الصحة العالمية ضد إستخدام العقار "رامديسيفير Remdesivir " في علاج مرضى كوفيد-19 .. كيف يفهم ذلك ؟
[5]. SARS-CoV-2 Second Wave & the continuous rise of infection in Algeria; How did that happen ? What to do ? .. brief suggestions !
الموجة الثانية لفيروس كورونا و الإرتفاع المستمر لحالات الإصابة في الجزائر؛ كيف حصل ذلك ؟ وماذا يمكن القيام به للمواجهة ؟ .. ملخص اقتراحات !
[6]. Why is Coronavirus, SARS-CoV-2, highly contagious ?
لماذا فيروس كورونا المستجد هو شديد العدوى ؟
[7]. Possible Molecular Basis behind the late surge of COVID-19 in Algeria.
الأسس الجزيئية المحتملة وراء الإرتفاع الأخير لحالات كوفيد-19 في الجزائر ..
[8]. Hydroxychloroquine based treatment protocols for COVIC-19 need reviewing .. a call from recent research.
بروتوكولات علاج كوفيد-19 المعتمدة على هيدروكسي-كلوروكوين في حاجة إلى المراجعة .. نداء من آخر البحوث في المجال. ء
[9]. النسخة β-v.2.0 لـ نظام 'Viruses' و " جينومات " فيروس كورونا .
The β-v.2.0 of 'Viruses' tool & the Genomes of SARS-CoV-2 .. . .. . .. . .. i
[10]. سلاسل الجينوم الكامل لفيروس كورونا SARS-CoV-2 المكتشف في ولاية البليدة، الجزائر ..
Algerian Complete Genome sequences of SARS-CoV-2 strains detected in Blida province .. . .. . .. . .. . i
[11]. تطبيق 'Viruses' مصدر للبيانات العلمية المدمجة حول فيروس كورونا المستجد ..
'Viruses' a new tool to finding scientific data on 2019-nCoV .. . .. . .. . .. . .. . .. . ..i
[12]. الجينوم الكامل لفيروس كورونا الجديد 2019-nCoV ..
The complete Genome of the new Coronavirus 2019-nCoV .. . .. . .. . .. . .. . i
[13]. التركيب الفراغي لـِ ماكنة تكاثر فيروس كورونا، إنزيم البلمرة RdRp وآمال في دواء لـ COVID-19 يستهدف هذا الإنزيم ..
Structure of the Replication Machinery of SARS-CoV-2, the RdRP enzyme and prospect for a drug targeting the enzyme. .. . .. . .. . .. . .. . .. . .. . i
[14]. مركب مُحسَّن لـِ ألفا-كيتوالأميد المثبط لإنزيم بروتياز من كورونا يوفر أملا في إيجاد دواء لـِ COVID-19 ..
Improved α-ketoamide inhibitor of SARS-CoV-2 main Protease gives hopes for a cure against COVID-19 . .. . .. . .. . .. . .. . .. . .. . .. . .. . .. . .. . .. . .. . .. . i
[15]. التركيب الفراغي لبروتين من غلاف فيروس كورونا الجديد .. وآمال في اللقاح والعلاج.
3D-structure of a Spike protein from the New Coronavirus .. . prospects for vaccines & cures .. . .. . i
[16]. التركيب الفراغي لإنزيم 'بروتياز' من فيروس كورونا الجديد ..
New Coronavirus Protease Structure .. . .. . .. . .. . .. .. . .. . .. . i
[17]. China approves first anti-viral drug against coronavirus Covid-19 ..
الصين تصادق على أول دواء مضاد لـ فيروس كورونا الجديد .. . .. . .. . .. . .. . .. . .. . .. . .. . .. ء
[18]. فيروس كورونا، ألإنسان و بحـر المعرفة ..
Coronavirus, Man & the ocean of Knowledge .. . .. . .. . .. . .. . .. . .. . .. . .. . .. i
[19]. منظمة الصحة العالمية تعلن حالة الطوارئ ذات أبعاد دولية في وصف حالة تفشي فيروس كورونا الجديد 2019-nCov بالصين ..
World Health Organization - Europe announces the new Coronavirus, 2019-nCoV, outbreak a Public Health Emergency of international Concern (PHEIC). .. i
أنظر كذلك:
أزكى تحيات العام الجديد .. . .. . .. . .. . .. . .. . .. . .. . .. . .. . .. . .. . .. . .. . .. . .. Best of the New Year regards
Author: A. Rachedi
E-mail: rachedi@bioinformaticstools.org
Date: - 10 January 2021 vr.1
- 31 January 2021 vr.2
E-mail: rachedi@bioinformaticstools.org
Date: - 10 January 2021 vr.1
- 31 January 2021 vr.2
Images: